Странные совпадения Как ученые открыли ультраконсервативные элементы в ДНК

В последовательностях ДНК самых разных животных - от рыб до человека - в середине прошлого десятилетия были найдены совершенно идентичные участки. С тех пор считалось, что это странное свойство высших позвоночных, однако недавно нечто подобное было найдено и у растений. Правда, для этого ученым понадобилось изобрести новый метод анализа последовательностей ДНК, который, как выясняется, может пригодиться и для других задач. О том, как это удалось, "Ленте.ру" рассказал один из авторов работы, американский ученый российского происхождения Дмитрий Коркин.
"Лента.ру": Расскажите, пожалуйста, о вашем открытии. Что вам удалось обнаружить?
Дмитрий Коркин: Мы пытаемся изучать абсолютно идентичные элементы в полной последовательности ДНК (геномах), разных организмов. У этих участков стопроцентная схожесть, с математической точки зрения, наверное, их можно называть тождественными.
В 2004 году Дэвид Хауслер и Джил Бежерано (Gill Bejerano & David Haussler), которые тогда работали в Калифорнийском университете Санта-Круз, опубликовали в журнале Science статью о результатах своих экспериментов. Они сравнили геномы мыши, крысы и человека и обнаружили, что в них существуют достаточно длинные (более 200 нуклеотидов) абсолютно идентичные участки.
Первая работа об этих ультраконсервативных участках появилась только в 2004 году? Но ведь сравнение геномов началось гораздо раньше. Геномика, биоинформатика к тому моменту давно уже этим занимались. Почему эти элементы не удавалось обнаружить раньше?
Их находили, но описывали как частные случаи. К тому времени были известны некоторые повторяющиеся элементы, образованные повторением достаточно маленьких нуклеотидных "слов".
Есть, например, теломеры, конечные участки хромосом. Известно, что на концах хромосом существуют последовательности, которые являются повторениями одного и того же "слова" сотни, а иногда и тысячи раз. Эти слова состоят всего из 7 "букв".
Однако полный анализ и идентификация сложных ультраконсервативных элементов, в которых нет никаких повторений, был впервые сделан Бежерано и Хауснером в 2004 году. Основная причина, по которой эту задачу не удавалось решить раньше, заключалась в сложности самого алгоритма сравнения целых геномов.
Что такое вообще ультраконсервативные участки? Чем они отличаются от участков ДНК, кодирующих важные гены, которые часто очень слабо различаются у разных организмов?
Геном - наследственная информация организма, заключенная в полной последовательности ДНК. У животных в геном включают и последовательность ДНК, содержащуюсяся в митохондриях, а у растений еще и в хлоропластах.
Ген - участок ДНК, последовательность которой кодирует последовательность одного белка. У высших организмов доля генома, занимаемая генами, существенно меньше доли некодирующих участков. (Раньше геном называли единицу наследственности, связанную с каким-то признаком, не обязательно кодированием белка, но это определение в целом устарело.)
Некодирующий участок - участок генома, не относящийся к генам.
Консервативный - если участок ДНК очень похож у разных видов, то его называют консервативным. Чем более далекие виды обнаруживают сходство в последовательности, тем более консервативным называется такой участок.
Ультраконсервативный - участок, совпадающий на 100 процентов. Без замен, удалений и вставок.
Выравнивание - расположение последовательностей одна под другой так, чтобы наибольшее число нуклеотидов в последовательности совпало. Выровнять можно любые две последовательности, но это не говорит об их родственности.
Отличаются они тем, что не допускают внутри себя никаких мутаций. Если произвести сравнение двух последовательностей, то на обычных консервативных участках мы будем замечать какие-то точечные мутации и различия все же будут.
Даже если это что-то очень консервативное, например гены рибосом?
Даже если это что-то очень консервативное, абсолютно. Повторюсь: частные случаи, когда у близких видов находили идентичные участки генома, были известны. Вы привели в пример гены рибосом. О них известно, что какие-то части этих генов были идентичны и не допускали никаких мутаций.
Бежеран и Хауслер впервые обнаружили, что у разных организмов существуют идентичные участки генома, которые не являются генами рибосом, не являются повторяющимися элементами и которые могут быть окружены участками хоть и сходными, консервативными, почти совпадающими, но при этом допускающими гораздо большее количество мутаций.
То есть, взяв такие почти совпадающие участки, мы можем посчитать в них частоту мутаций. Потом, учитывая эту частоту, можно прикинуть, с какой вероятностью обнаруженный Бежерано с Хауслером ультраконсервативный участок длиной 200 нуклеотидов будет лишен мутаций вследствие случайного совпадения. Так вот, эта вероятность оказывается ничтожно мала. Следовательно, это не просто консервативный, а ультраконсервативный участок.
Помимо мыши, крысы и человека ультраконсервативные участки потом были обнаружены и у других организмов?
Да, совершенно верно. Потом эти участки начали находить в геномах других животных. И насколько я знаю, сумели довести этот поиск до рыб. Но они уже были не у всех рыб. У нескольких рыб нашли последние отголоски этих ультраконсервативных элементов. Поэтому было сделано предположение, что ультраконсерватизм, сам механизм как таковой, это достаточно поздний механизм, и он существует только у позвоночных, даже не просто у позвоночных, а у тетрапод, то есть у четвероногих животных.
Но потом выяснилось, что это не совсем так?
Да. Я тогда еще был постдоком в Калифорнийском университете в Сан-Франциско, и к нам с докладом как раз приехал Бежерано. Это был 2006 или 2007 год. И у меня возник естественный вопрос: может быть, такой же механизм есть у растений? Потому что, в принципе, они похожи с эволюционной точки зрения. В том смысле, как высшие растения и животные разветвились. Но выяснилось, что сделать это для растений гораздо более сложно.
Это, видимо, связано с особенностями геномов растений?
Да, за счет того, что кусочки, на которых можно выравнивать геномы растений, очень маленькие.
Если вы помните, была такая работа, где хромосомы человека покрасили в разные цвета, и потом эти участки отобразили на геном мыши. Оказалось, что эти участки присутствуют в геноме мыши, но не в виде целых хромосом. Соответствующие участки хромосом человека и мыши оказались сильно перемешаны.
Поэтому необходимо было не просто целиком взять геном и сравнить, выровнять попарно хромосому человека с хромосомой мыши, а нужно было это делать обязательно по кускам. Эта задача гораздо более сложная, но она оказалась решаема, потому что куски, участки генома человека, мыши и крысы (потом оказалось, и у многих других животных), достаточно длинные. Это позволило использовать метод выравнивания для решения такой сложной задачи.
Как и в примере между мышью и человеком, у растений эти кусочки мельче и перемешаны они сильнее. По сравнению с размером самих геномов они составляют доли процента.
То есть вы первые, кому удалось найти такие последовательности у растений?
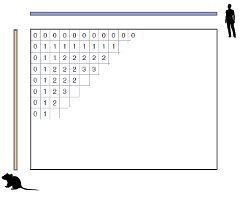
Иллюстрация заполнения матрицы при работе алгоритма выравнивания. Сравниваются геном человека и мыши.
(Нажмите, чтобы увеличить)
Lenta.ru
Совершенно верно. Мы занялись этой задачей буквально через полгода после того доклада, я получил должность в университете Миссури, и там мы сразу же начали разрабатывать методы для решения этой задачи. На самом деле при разработке нового алгоритма попутно появились совершенно другие направления, которые используются теперь для анализа, например, белковых последовательностей.
Оказалось, что с математической точки зрения задача сама по себе очень тяжелая. Сначала возникло понимание того, что все-таки нужно эту задачу решать не методами выравнивания, а как-то по-другому. Но тут, к счастью, помогло мое образование как математика, я научную степень получал в Канаде по компьютерным наукам, и мы вместе с профессором с кафедры компьютерных наук в университете Миссури создали алгоритм, в котором при поиске идентичных последовательностей не используется выравнивание, а используется так называемый "Hash-mapping".
Прежде всего нужно пояснить, как работает классический алгоритм выравнивания. Он состоит в том, что сначала строится огромная таблица-матрица. При сравнении двух геномов эта матрица двумерная, при сравнении трех - трехмерная и так далее. В ячейках такой матрицы мы ставим совпадения или несовпадения соответствующих нуклеотидов последовательности - A, T, G и С. Мы заполняем матрицу, в ячейках у нас, условно говоря, 1 - это совпадение, 0 - это несовпадение. Только мы не просто 1 и 0 ставим, мы прибавляем 1 или 0. То есть каждый раз, когда мы увеличиваем длину последовательности, которую мы анализируем, мы будем прибавлять либо 1, если следующая позиция совпала, либо 0, если следующая позиция не совпала.
То есть если у нас матрица четыре на четыре, четыре буквы на четыре буквы, то у нас в правой нижней ячейке будет стоять цифра 4, если все совпало.
Да, если все совпало,
Если следующая, пятая позиция не совпадает, в ней снова будет стоять 4, хотя было бы 5, если бы последовательности совпали?
Совершенно верно. И теперь, имея такую таблицу, мы ищем путь из правого нижнего угла матрицы в левый верхний угол. Мы ищем такой путь, который проходил бы через максимальные значения. И этот путь фактически будет одним из решений этого выравнивания, хотя решений может быть много.
Так работал этот алгоритм по поиску ультраконсервативных участков у животных. Вы обнаружили, что он совершенно неприменим к растениям? Ваш алгоритм работает как-то по-другому?
Да. Идея алгоритма, который решили применить мы, заключается в том, что вместо того чтобы выравнивать строки, мы просто-напросто в каждой из последовательностей, ставим… представьте себе маленькие флажки разных цветов. Каждый флажок одного цвета соответствует, скажем так, слову из 8 букв. Красный, например, флажок, соответствует слову AAAAAAAА, синий соответствует слову АТATATAT, и так далее. Что наш алгоритм делает? Он шагает каждые, скажем, 50 нуклеотидов и через каждые 50 нуклеотидов ставит такой флажок и окрашивает его в определенный цвет, в зависимости от того, какое слово ему встретилось.
При этом "слово" имеет длину 8 "букв", а шагает он через 50?
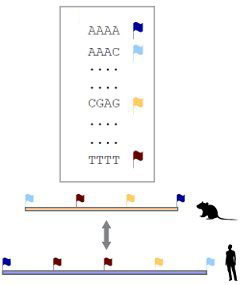
Иллюстрация алгоритма работы хешмеппинга.
(Нажмите, чтобы увеличить)
Lenta.ru
Через 50, да. Теперь, когда мы сравниваем геномы, на самом деле мы смотрим не на геномы, а на набор этих флажков. Когда две последовательности идентичны, они начинаются и кончаются на флажки одинакового цвета. То, что не одного цвета, можно даже не рассматривать. Это очень грубая идея, но это основа хэшмеппинга.
И первое, что мы сделали - мы конечно же, проверили свой метод на уже найденных ультраконсервативных элементах. Ключевая проверка заключалась в том, сможем ли мы с помощью нашего метода обнаружить те элементы, которые нашли Бежерано и Хаустлер.
Что получилось?
Мы не только обнаружили этот 481 элемент, мы нашли новые элементы в тех же самых данных, которые они анализировали. Мы нашли 12 новых элементов. Причем часть из них была в родственных, но очень сильно различающихся, а часть - вообще в неродственных (несинтеничных) участках генома, поэтому исходный алгоритм просто не мог бы их обнаружить.
То есть ваш алгоритм был применим не только для растений, он еще и оказался более удачным для животных? Что вы обнаружили, когда сравнили геномы растений? Что это были за ультраконсервативные элементы?
Чем больше мы в них копались, тем больше выяснялось, что у них очень мало общего с элементами животных геномов. Их было существенно меньше по общему числу, но гораздо больше по количеству типов.
Сколько вот эти 6 растений между собой содержали больших идентичных участков?
Мы сравнивали шесть видов растений: арабидопсис, или резуховидку (Arabidópsis thaliána), сою, рис, хлопковое дерево, сахарный тростник и виноград. Все шесть растений вместе идентичных последовательностей содержали совсем немного. Там буквально меньше десятка было. Если рассматривать тройки, консервативных элементов было гораздо больше. Выяснилось, что гораздо больше, чем у животных (в процентном отношении), было повторяющихся элементов.
Известно, что у родственных организмов родственные гены обычно не совпадают, поскольку со временем накапливают мутации. Мы знаем о том, что есть более консервативные гены, мутации в которых часто приводят к дефектам и быстро отбраковываются отбором, и есть менее консервативные гены, в них накапливается больше мутаций. Мы знаем, что и внутри самих генов есть участки, которые более или менее консервативны, чем другие (например, кодирующие активный центр фермента). Но никогда совпадение не бывает тождественным (если только не у очень близких видов). Всегда наблюдаются какие-то ошибки. Вы же прогнали через алгоритмы сравнения целые геномы совершенно разных организмов и увидели какие-то удивительные полностью совпадающие участки. Что вообще это такое? Эти участки выполняют какую-то функцию или это просто какое-то недоразумение?
Это называют здесь "вопрос на миллион долларов".
Потому что это первый вопрос, который возникает у любого интересующегося человека?
Конечно. Начнем с того, что вы сказали о том, что ожидаете консервативности именно в генах, то есть в участках ДНК, кодирующих белки. Первое же, с чем столкнулись еще Бежерано и Хауслер, заключалось в том, что очень много из обнаруженных ультраконсервативных элементов являются вообще некодирующими участками ДНК, то есть тем, что раньше называли "мусорная ДНК". Они не имели отношения к кодированию последовательности белков.
И функция ультраконсервативных элементов до сих пор неизвестна. На самом деле, всего лишь один эксперимент был сделан, где один из элементов был удален - у мыши, и выяснилось, что мышь получилась абсолютно живая. Это, опять-таки, повергло в шок исследователей, потому что этому не было объяснений. Я бы сказал так, что на данный момент эти открытия дают гораздо больше новых загадок, нежели ответов.
Подождите, мы обнаружили у мыши какой-то участок, который совпадает с таким же участком у человека. Удалили этот участок, и мышь себя чувствует прекрасно. Может быть, просто в оба генома встроился какой-то вирус, который не имеет никакой функциональной роли? Откуда мы знаем, что эти участки действительно сохраняются миллионы лет, а не являются следствием недавней вставки? Где доказательство того, что эти участки сохранялись все эти миллионы лет?
Картина у животных и у растений отличается. В нашей статье мы показали, что как минимум часть совпадающих участков у растений действительно появились за счет механизмов вставки, только не вирусов, а участков митохондриальной ДНК в ядерную ДНК. И это происходит у растений постоянно и с более-менее одними и теми же участками митохондриальной ДНК. Объяснить, почему это происходит с теми или иными участками, мы пока не можем.
У животных все по-другому, но как именно, очень сложно сказать. Если бы это было последствие работы вируса, то сложно представить себе вирус, который вставляет в геномы разных видов с завидной постоянностью одни и те же участки. Особенно, если брать во внимание тот факт, что эти участки все-таки находятся в родственных регионах генома.
То есть, навряд ли вирус вставлял бы разным организмам свою ДНК в одно и то же место генома?
Один и тот же участок в одно и то же окружение, да.
Интересно, но удивительно мало понятно в данном случае, что происходит и какое это будет иметь значение.
Да, и самое интересное: 8 лет прошло со дня открытия, а на самом деле, не так-то много и понятно.